杭州医学所等发表全球最大规模结直肠癌多组学研究成果
| 来源: 杭州医学研究所 【字号:大 中 小】
8月7日,中国科学院杭州医学研究所组学与智慧医疗中心联合瑞典乌普萨拉大学、华大生命科学研究院、华大基因智惠医学研究院在《自然》(Nature)上,发表了题为Prognostic genome and transcriptome signatures in colorectal cancers的研究论文。研究公布了全球首个大规模结直肠癌多组学的研究结果,并结合临床信息系统总结了与临床结果和疾病进展密切相关的分子特征。
全球范围内结直肠癌每年新增病例约190万例,发病率居所有癌症第三位,大约20%的患者在确诊时已发生转移,其死亡率更是高居各类癌症第二位,是严重威胁人类生命健康的重大疾病之一。
为了加深对结直肠癌发病机制的理解,识别驱动事件并确定其预后特征,科研团队针对瑞典U-CAN队列中结直肠癌样本进行了测序分析。基于华大智造DNBSEQ测序平台,研究团队对1063名结直肠癌患者的样本进行了全基因组和转录组测序,包括782例结肠癌和281例直肠癌,21%(223例)是微卫星不稳定(MSI)肿瘤,总共生成近412T数据,其中94%的病人有完整的5年临床随访记录,形成目前全球最大的结直肠癌多组学研究。此研究系统绘制了与结直肠癌相关的基因组变异事件全景,联合基因表达分型揭示了与预后密切相关的分子特征。
根据全基因组的肿瘤突变负荷高低,患者被分为超突变型(23%,HM)和非超突变型(77%,nHM)。由于MSI肿瘤通常因为DNA错配修复基因缺陷而表现出超突变特征,此队列中超90%的HM肿瘤为MSI。具有HM特征的微卫星稳定(MSS)肿瘤则携带POLE或其他DNA修复通路相关基因突变。APC, TP53和KRAS等驱动基因在MSS肿瘤突变频率更高,而PIK3CA及BRAF等的突变则在MSI肿瘤中更为富集。基于该队列的基因组突变共鉴定到96个显著突变基因,其中24个为潜在的新驱动基因,其中WNT, EGFR和TGF-β通路相关的驱动基因与结直肠癌生存显著相关。
研究团队从突变特征谱分析鉴定出47个已知的和9个新发现的结直肠癌突变特征(SBS-CRC1、SBS-CRC2、DBS-CRC1、DBS-CRC2、DBS-CRC3、DBS-CRC4、DBS-CRC5、ID-CRC1、ID-CRC2),其中SBS28和DBS-CRC5与POLE突变的MSS肿瘤显著相关,SBS-CRC1、DBS-CRC3和ID-CRC1是DNA错配修复通路缺失的突变特征,而SBS44阳性的HM样本具有更长的生存期。新的突变特征将有可能成为结直肠癌早期诊断和预后评估的生物标志物,为结直肠癌的预防和治疗提供新的思路。
通过时序分析研究团队推导出9个与癌症发生早期事件相关的驱动突变基因(APC、TP53、KRAS、BRAF、ZFP36L2、TCF7L2、FBXW7、BCL9L和SOX9),而TRPS1、GNAS和CEP170这3个基因的突变则倾向于在癌症发生的后期阶段出现,这些发现为结直肠癌的早期检测和靶向治疗的开发提供了临床研究策略,并揭示了与肿瘤后期侵袭和转移相关的重要分子变化。此外,利用高深度全基因组测序的优势,本研究在线粒体基因组和非编码区域中也发现了与疾病相关的突变,并对与疾病进展显著相关的突变进行了系统性的总结,这些突破性的发现将进一步推动对结直肠癌发病机制的理解。
研究团队利用同一肿瘤样本的基因组和转录组数据对突变基因和基因表达水平进行了整合分析,验证了RTK-RAS、PI3K、P53和TGF-β通路相关基因EGFR、KRAS、PIK3CA、CDKN2A、TGFBR1和ACVR2A的突变与基因表达增加有关,而无论是否为MSI肿瘤,其抑癌基因APC、PTEN和TP53上的突变均导致基因表达降低。
研究团队进一步基于肿瘤基因表达差异谱解析出5个具有不同分子特征的预后亚型(CRPS),相较于结直肠癌经典的共识分子分型CMS(CMS),利用独立队列转录组数据构建的CRPS能更加准确地预测预后,并在不同结直肠癌队列中得到验证。值得注意的是,在经典的CMS分型中,CMS4间质型肿瘤具有基质细胞浸润较高、TGF-β通路激活等特征,且被普遍认为是预后较差的肿瘤,但在该研究新构建的CRPS分型系统中,部分CMS4型肿瘤实际上被判定为预后较好的CRPS2型,而CRPS2型肿瘤具有上皮细胞特征,且在基因组20q11区域具有较多的扩增,这些结果提示整合基因组和转录组数据的分子分型,能得到更精细准确的患者预后分层,对优化临床肿瘤分型,指导结直肠癌精准治疗具有重要意义。
研究团队基于深度学习算法ResNet50开发的CRPS分型工具已在Github开源,为结直肠癌后续的研究和治疗提供更加精准的分子分型,为预后评估和深入理解疾病机制提供了重要信息和方法。
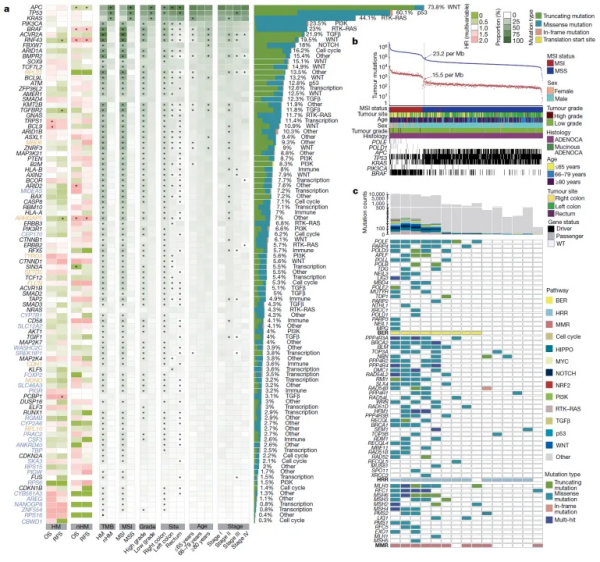
图1. 结直肠癌驱动基因的全景描绘
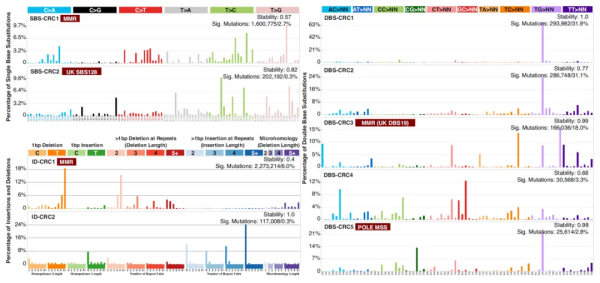
图2. 新发现的结直肠癌突变特征
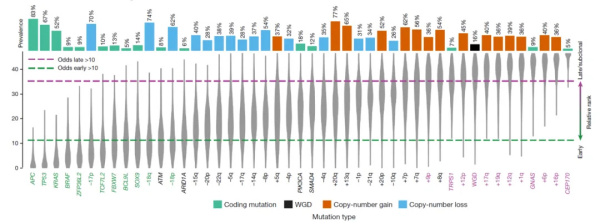
图3. 基于驱动基因及结构变异的时序分析
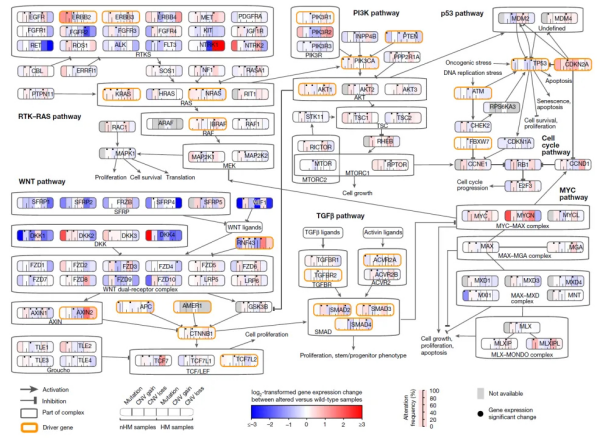
图4. 结直肠癌相关通路中的基因突变和基因表达水平的综合分析
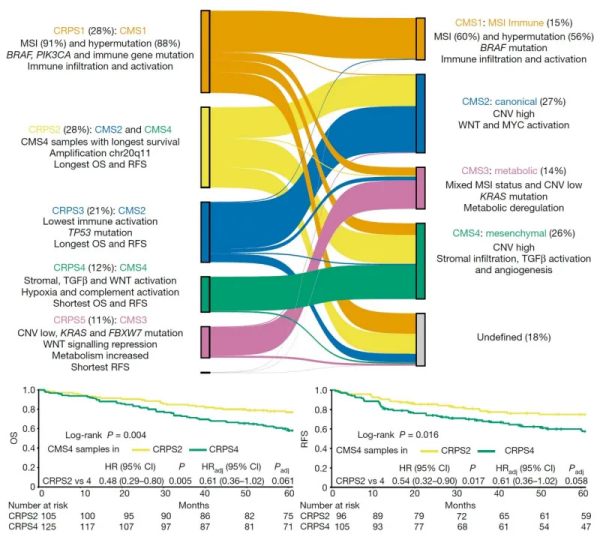
图5. CRPS分型系统中各个亚型的分子特征及预后表现
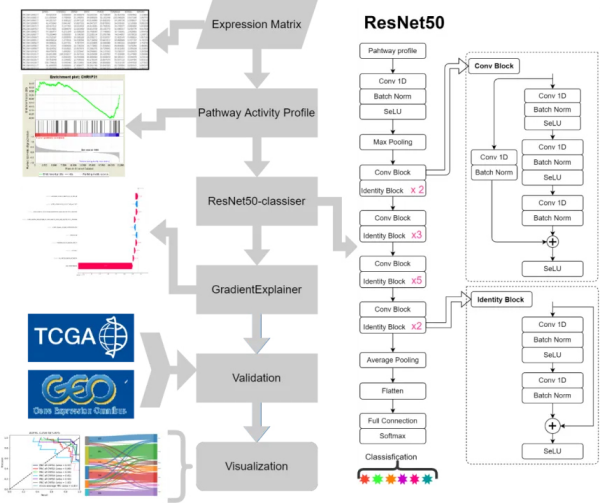
图6. 基于深度学习的CRPS分型工具
