研究揭示古菌因子依赖型转录终止分子机制
| 来源: 上海免疫与感染研究所 【字号:大 中 小】
9月25日,中国科学院上海免疫与感染研究所王程远研究组联合美国罗格斯大学Richard Ebright团队、美国科罗拉多州立大学Thomas Santangelo团队,在《自然》(Nature)上发表了题为Structural basis of archaeal FttA-dependent transcription termination的研究论文。该研究解析了古菌FttA因子依赖型转录终止复合物的三维结构,揭示了FttA因子介导古菌RNA聚合酶转录终止的分子机制。
转录终止是RNA聚合酶停止转录延伸,并使RNA从DNA上解离释放的过程。异常的转录终止会干扰下游基因的表达,阻碍RNA聚合酶的循环利用,破坏基因组的稳定性。因子依赖型转录终止是普遍存在于细菌、古菌和真核生物中的一类转录终止机制。近年来,细菌和真核生物的转录终止的分子机制相继被阐明。然而,作为第三生命域的古菌,它的因子依赖型转录终止的机制尚不清楚。
古菌转录终止因子FttA又称aCPSF1。FttA是真核生物整合子复合物中INTS11亚基和切割与加聚腺苷酸特异性因子复合物中CPSF73亚基的同源蛋白,负责古菌中多数转录单元的转录终止。FttA具有核糖核酸内切酶活性和5'→3'核糖核酸外切酶活性。同时,唯一在三域内保守的转录因子Spt5能够促进FttA的转录终止活性。然而,FttA如何介导古菌RNA聚合酶的转录终止以及Spt5如何促进转录终止的机制尚不清楚。
该研究利用体外RNA切割和解离实验,确认了各个组分可以组装成具有转录终止活性的复合物。由于转录终止的过程动态且不稳定,为了捕捉稳定的转录终止复合物,研究使用突变的FttA蛋白进行复合物组装,并通过单颗粒冷冻电镜方法解析了分辨率为2.2Å-2.5Å的转录终止复合物结构。结构表明,FttA结合在RNA退出通道的外侧,使RNA可以直接从RNA聚合酶退出通道而进入FttA的切割活性中心。Spt5在FttA与转录延伸复合物(TEC)之间发挥桥梁作用,其N端结构域结合RNA聚合酶,C端结构域结合FttA,解释了Spt5如何促进FttA介导的转录终止。FttA以二聚体形式发挥作用,而FttAprox和FttAdist在功能上不同。FttAprox负责执行终止所需的RNA切割,而FttAdist提供支持,通过扩展与mRNA的相互作用面,增强序列U的选择性和终止复合物的稳定性。FttA的二聚体形式增强了其RNA切割活性。FttA利用核糖核酸内切酶活性对mRNA进行切割,进而利用外切酶活性进行5'→3'切割mRNA,并沿着mRNA进行5'→3'转位,从而对TEC施加机械力,引发转录终止。
进一步,该研究比较了细菌、古菌和真核生物的因子依赖型转录终止机制。结果表明:在细菌中,具有转位酶活性的Rho因子结合RNA,并沿RNA的5'→3'方向转位,对TEC施加机械力,从而引发终止;在古菌中,具有核糖核酸内切酶和外切酶活性的FttA切割RNA,并同样进行5'→3'转位,触发终止;在真核生物中,INTS11或CPSF73切割RNA,随后INTS11、CPSF73或XRN2沿着RNA 5'→3'方向进一步切割和转位,引发转录终止。这些终止机制依赖于终止因子在RNA通道外侧与RNA聚合酶和RNA结合,通过转位酶活性或外切酶活性对TEC施加机械力,引发转录终止。该研究提出的三域生物因子依赖型转录终止机制的趋同进化模型,为探索生命的起源与进化奠定了研究基础。
该研究通过冷冻电镜解析了古菌FttA依赖性转录终止复合物的三维结构,阐明了Spt5蛋白介导FttA与RNA聚合酶的相互作用方式,揭示了细菌、古菌和真核生物中因子依赖性终止机制的基本统一性,并为进一步探究古菌转录终止及古菌转录-翻译偶联质量控制的结构和功能奠定了基础。
研究工作得到国家自然科学基金面上项目、上海市2023年度“科技创新行动计划”基础研究项目等的支持。
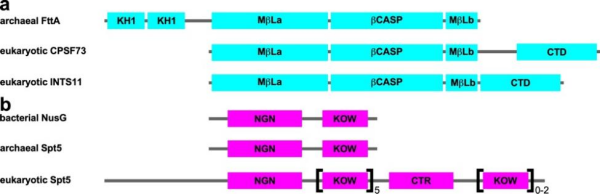
转录终止因子FttA和转录延伸因子NusG/Spt5
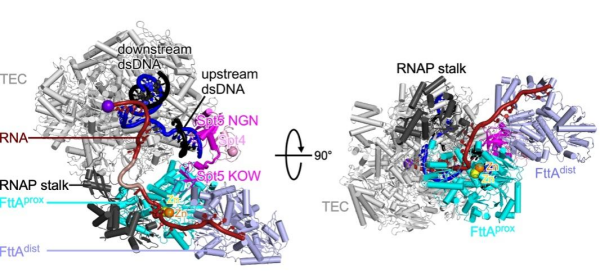
转录终止复合物结构
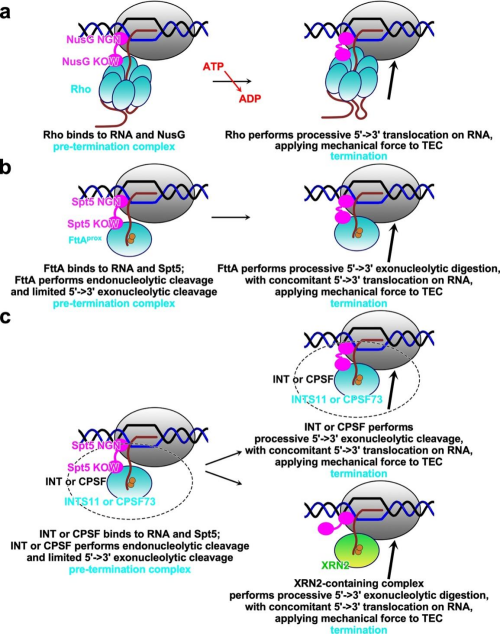
细菌、古菌和真核生物因子依赖性转录终止机制模型
