科学家开发出从空间组学数据解析肿瘤微环境的异质图学习方法
| 来源: 【字号:大 中 小】
6月13日,中国科学院分子细胞科学卓越创新中心陈洛南研究组与东华大学左春满团队合作,在《自然-通讯》(Nature Communications)上,在线发表了题为Dissecting tumor microenvironment from spatially resolved transcriptomics data by heterogeneous graph learning的研究成果。该研究提出了从空间组学数据解析肿瘤微环境异质性的深度学习方法(stKeep)——基于异质图模型集成空间转录的多模态数据(基因表达、物理位置、病理学信息、肿瘤区域)和分子网络信息(蛋白互作网络、基因调控网络和配体-受体互作网络),识别肿瘤微环境的异质性细胞状态、特异性基因-基因互作网络以及与疾病进展相关的细胞通讯模式。
癌症可视是一个肿瘤生态系统。其中,肿瘤细胞与其微环境细胞(包括免疫和基质细胞)协作,在各种恶劣环境如缺氧中挣扎求生。近期,陈洛南研究组与左春满团队发布了解析肿瘤内部异质性规律的工具——stMVC。而如何有效利用空间转录组数据揭示调控肿瘤异质性的内部分子互作和外部细胞通讯网络,是下一步的挑战。
该研究提出了名为stKeep的新模型。该模型构建了三种不同节点即细胞/spot、基因和肿瘤区域以及八种连接关系的异构图模型,用于刻画肿瘤微环境。进一步,研究采用基于注意力机制的多层关系图嵌入和对比学习算法,整合相关基因、肿瘤区域以及语义相连细胞的信息,计算细胞模块以检测更精细的细胞状态。类似地,通过整合相关细胞、细胞状态以及基因-基因关系的信息,计算基因模块以识别细胞状态特异的基因模块。与传统的细胞通讯方法不同,stKeep利用注意力机制来量化单细胞/spot通讯强度,并结合对比学习技术,推断反映肿瘤微环境内细胞状态差异的细胞通讯,从而计算与疾病状态相关的配体-受体对。
在三阴性乳腺癌的应用中,相较于其他方法,stKeep能够检测到肿瘤区域内更多的细胞状态。此外,通过整合配对的scRNA-seq数据,stKeep发现了一群此前研究被误分类为正常细胞的肌上皮细胞而实际上是肿瘤细胞,并推断这些细胞是促使疾病恶化的关键转录因子、配体和受体。
在结肠癌肝转移癌的应用中,stKeep识别了与结肠癌细胞转移到正常肝脏相关的关键细胞群体和细胞通讯机制。这些生物学发现进一步通过独立样本和临床数据得到了验证。
stKeep通过整合多模态和分子网络数据,构建细胞模块、基因模块和细胞通讯模块,从而解析肿瘤生态系统的异质性。同时,stKeep将为空间转录数据在临床预后和免疫治疗中的应用提供支持。
研究工作得到国家自然科学基金、国家重点研发计划、中国科学院战略性先导科技专项等的资助,并获得分子细胞卓越中心高性能计算平台的支持。
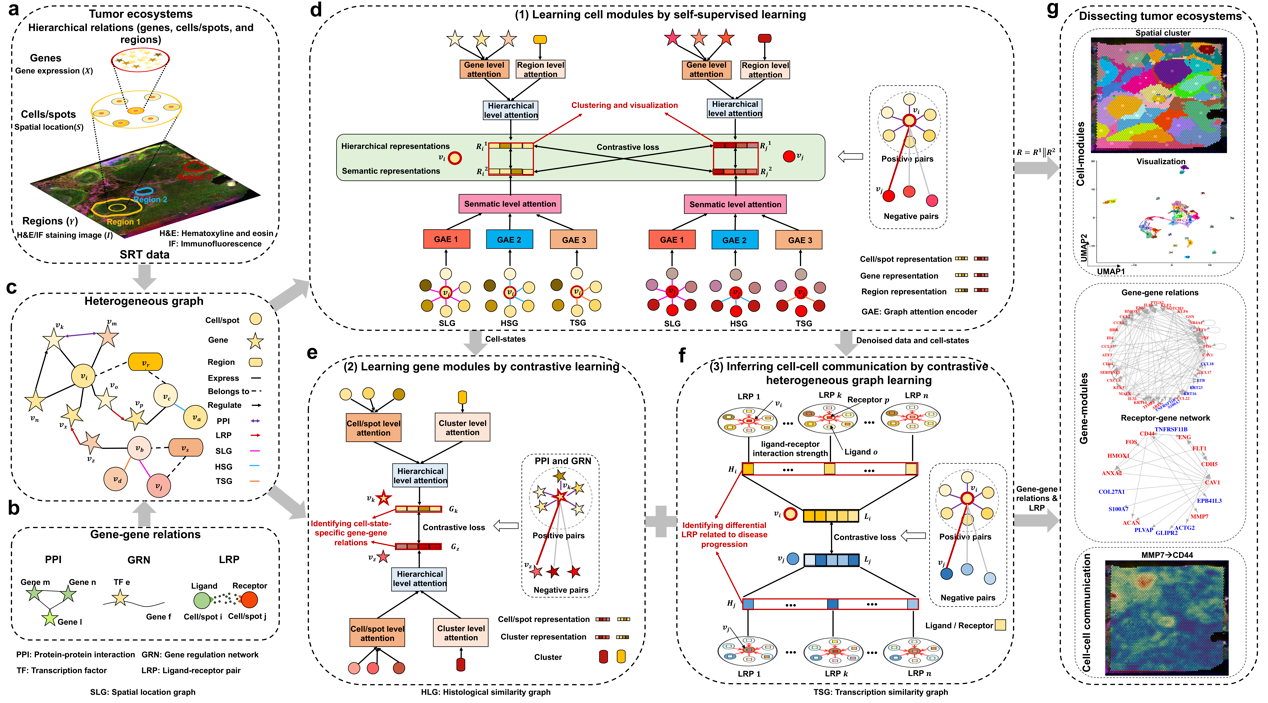
stKeep算法的框架
