北京基因组所等开发基于深度学习模型的空间转录组精细分辨率细胞注释算法
| 来源: 【字号:大 中 小】
细胞在组织和器官内的空间位置与其功能相关。新兴的空间转录组技术能够在组织中原位捕获转录信息,为解析基因表达谱从而解码细胞空间分布、破译组织的空间结构、探讨复杂的生物学功能提供了条件。
同时,现有的计算方法通过反卷积等方式实现了捕获域内混合细胞类型注释,但未对捕获域间未测量区域细胞类型进行填补,且分辨率提升空间有限。现有方法聚焦于基因表达信息,但未结合形态学信息进行细胞注释,且注释准确度有待提高。
近日,中国科学院北京基因组研究所(国家生物信息中心)杨运桂团队和数学与系统科学研究院张世华团队合作,在《基因组生物学》(Genome Biology)上发表了题为STASCAN deciphers fine-resolution cell-distribution maps in spatial transcriptomics by deep learning的研究论文。该团队针对不同空间转录组技术、不同生物组织建立了解码细胞空间精细分布、解析特异组织结构的新工具——STASCAN。
STASCAN基于深度学习模型,整合空间转录组基因表达数据和组织学图像,实现组织切片未测量区域细胞类型预测及测量区域捕获域内细胞细分注释。STASCAN的优势在于能够以高精度进行细胞类型注释并增强细胞空间分辨率,解析原始分辨率下无法识别的精细组织结构,进一步揭示细胞类型小生境,加速研究人员对发育和疾病的认知。同时,STASCAN仅基于相邻切片的组织学图像生成细胞分布图,从而能够构建更详细的3D细胞图谱,并减少实验成本。
STASCAN在公共涡虫空间数据集中进行原始分辨率下的基准测试,优于其他算法的精准细胞注释,能够仅从图像预测细胞类型。进一步,STASCAN可以在涡虫数据集中应用3个功能模块,揭示原始分辨率下无法识别的精细组织结构,定位细胞混合物的组成及其在亚分辨率下的不同位置,仅基于邻近染色图像实现细胞分布预测,构建超原始分辨率6倍的精细细胞三维分布图谱。
STASCAN可以应用于不同空间转录组学技术的跨物种的多个数据集。STASCAN在人类肠道数据集中定位肠道组织不同细胞层的边界;在人类肺数据集中特异识别微米级精细结构;在人类心肌数据集中预测测序缺失区域中的潜在细胞分布,重绘人体心肌梗死组织中的空间结构变化;在小鼠胚胎脑数据集中解码细胞空间精细分布,复刻小鼠脑发育过程中组织解剖学结构。
STASCAN提供了用于整合空间基因表达信息和组织学图像进行精细分辨率细胞注释的工具,在解码细胞空间精细分布和解析特异组织结构方面具有优势。随着空间转录组技术的发展和数据的积累,STASCAN有望为大规模空间转录组数据的整合分析提供支持。
研究工作得到国家重点研发计划和国家自然科学基金等的支持。
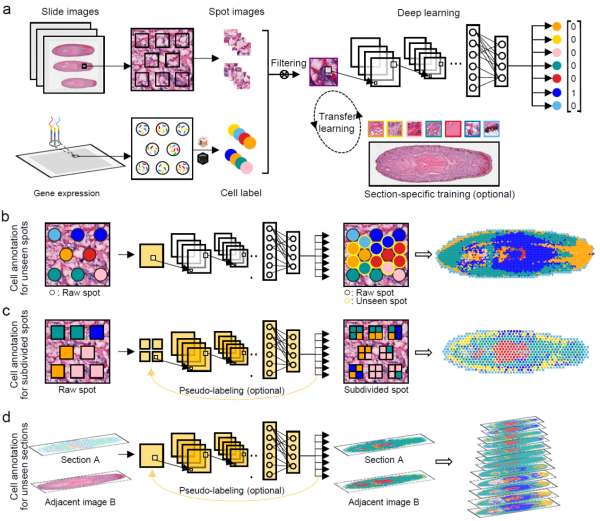
STASCAN模型框架图
